FreeSurferのファイルとBrainVISAのファイルの変換
FreeSurferのsurfファイルをGIfTIファイルに変換
GIfTIファイルをFreeSurferのsurfファイル変換
FreeSurferのmri_convertやmri_convertを使います。前者は三次元配列、後者はポリゴンメッシュデータです。
但し、mri_convert後は座標変換をする必要があります。
MGZファイルからNIfTIファイルへ変換
mri_convert
T1.mgz T1.nii.gz
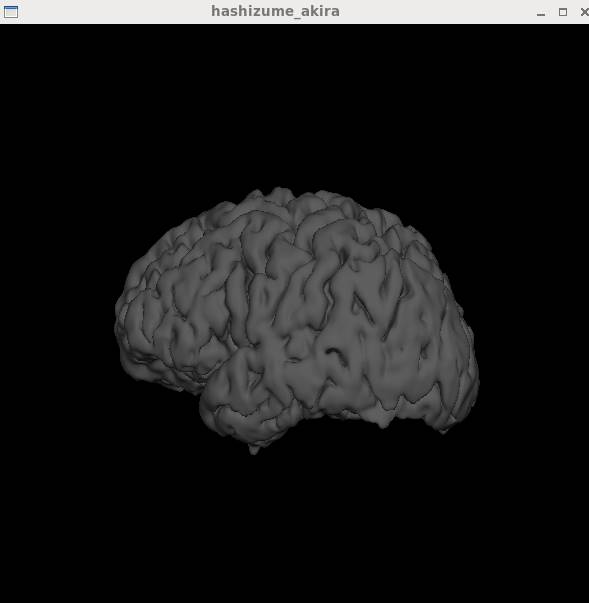
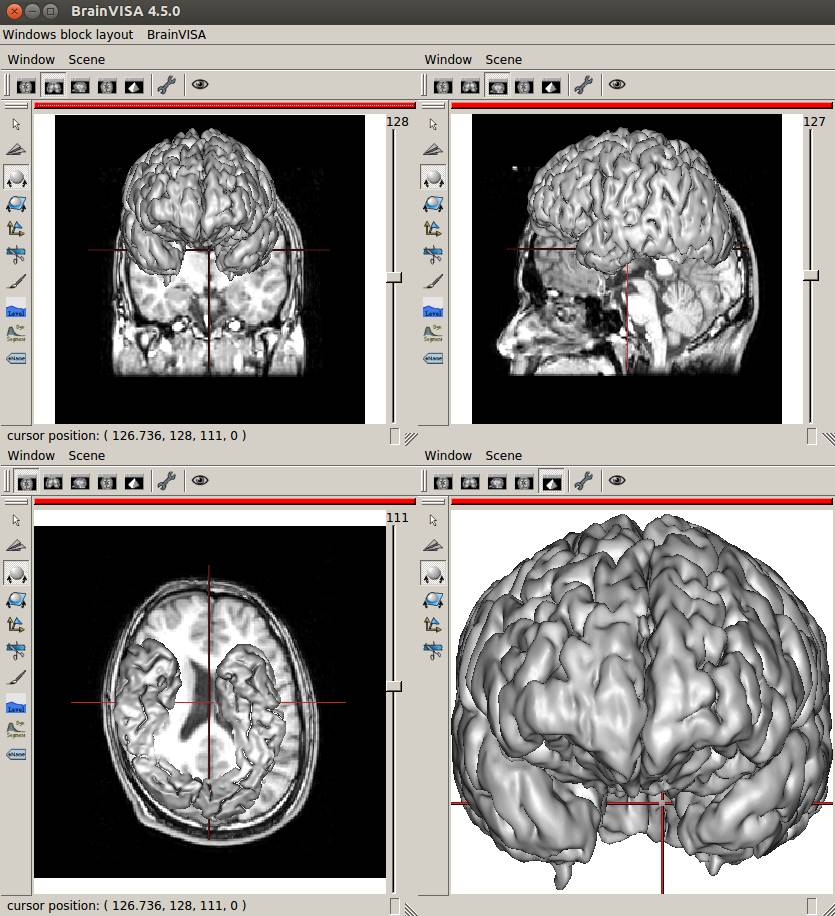
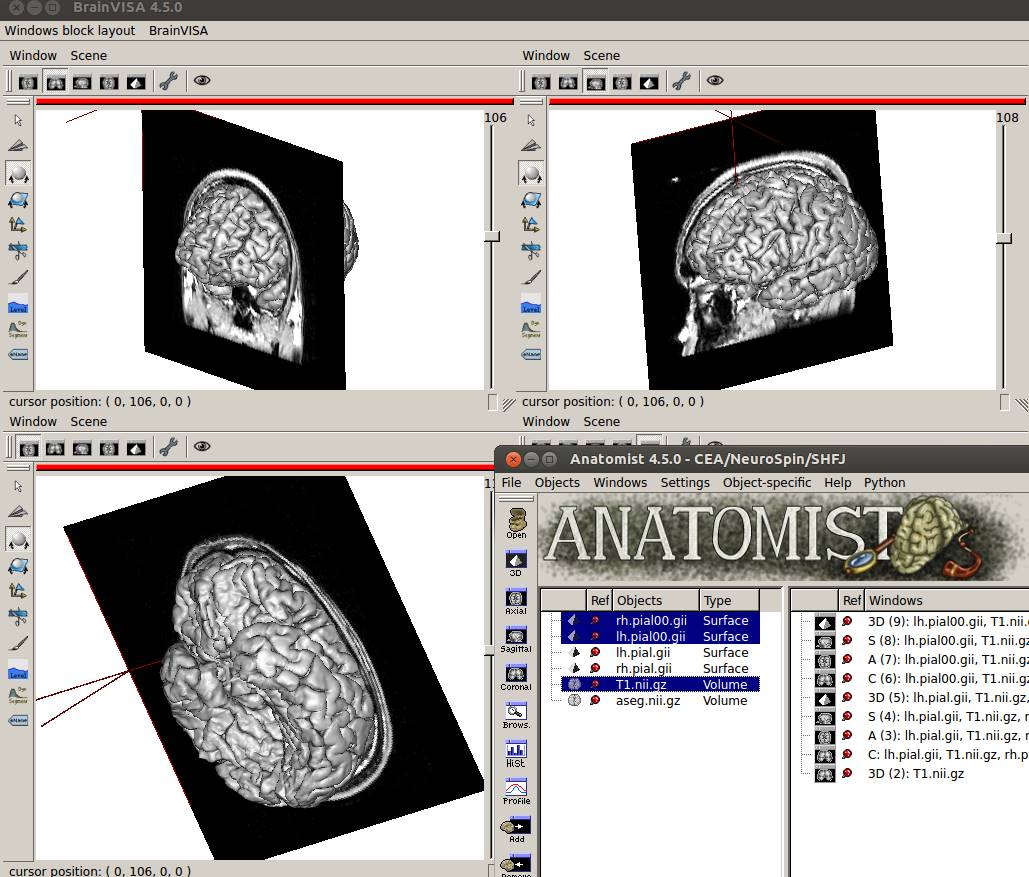
Anatomistで開いたところです。
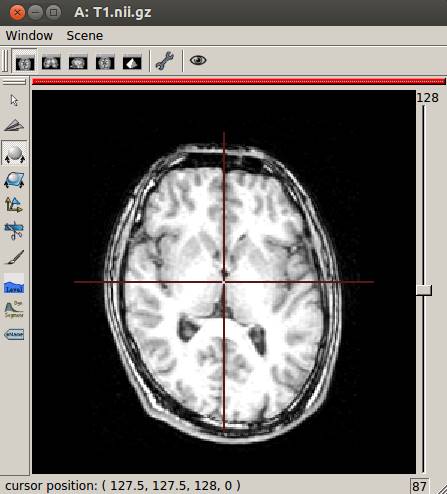
Windows→Open a 4 views blockをクリックします。
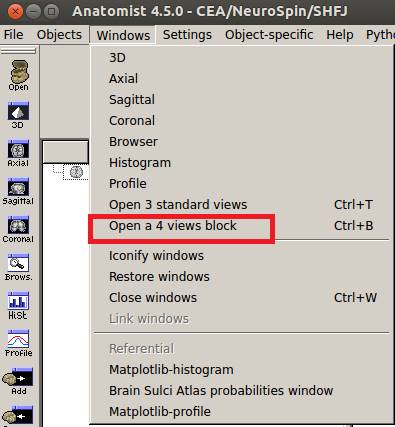
3断面で表示させてみました。
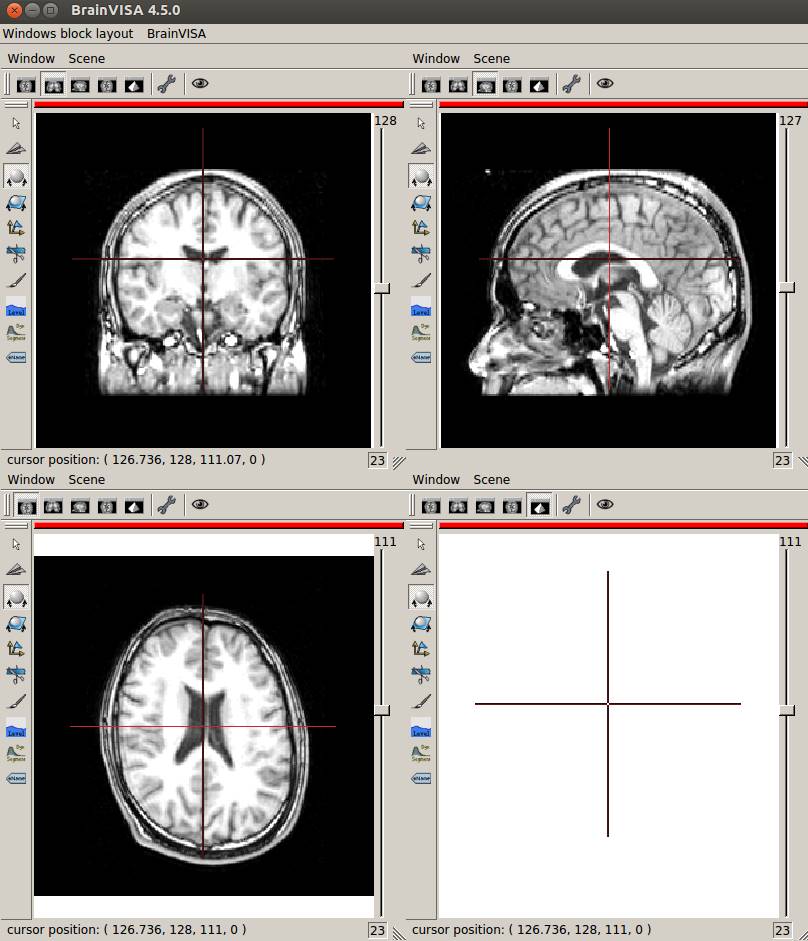
aseg.mgzをaseg.nii.gzに変換して四面Anatomistで見てみました。
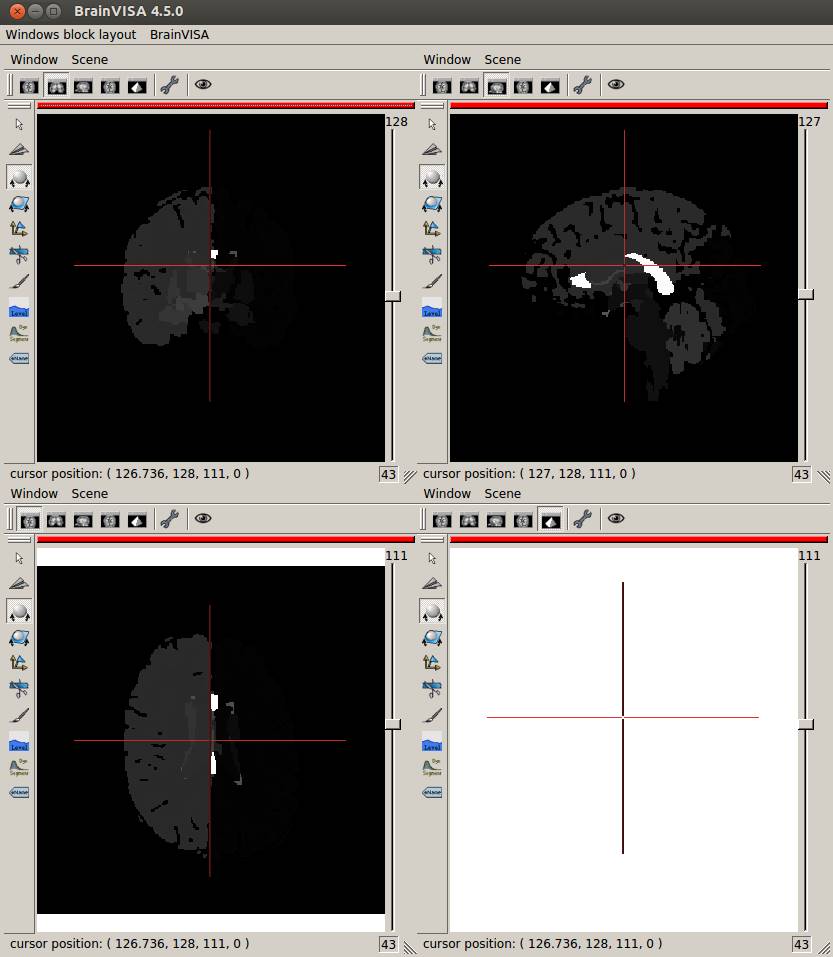
残念ながらBrainVISAで作成した脳表メッシュとは座標が異なるようです。
NIfTIファイルからMGZファイルへの変換
mri_convert
20000406_132343s002a1001.nii.gz 20000406_132343s002a1001.mgz
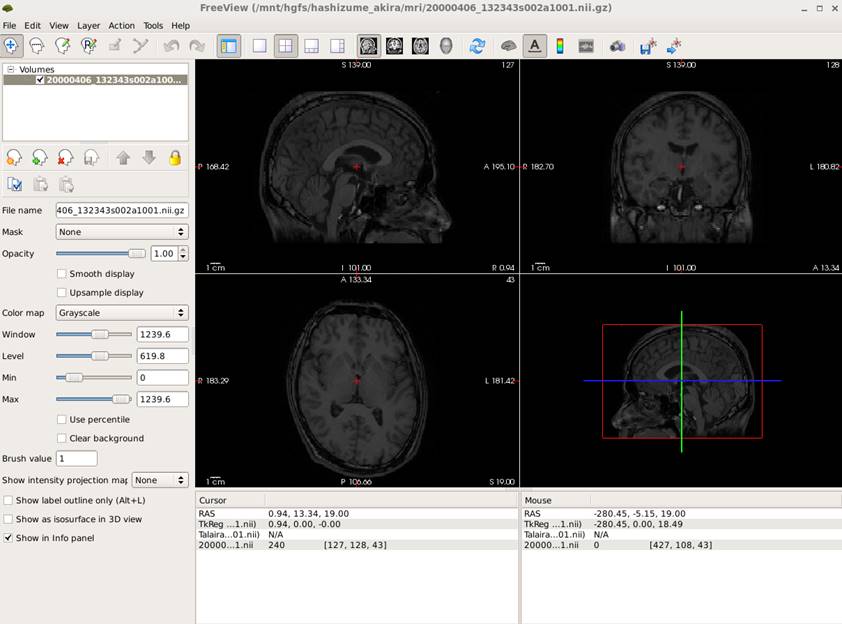
freeviewで開いたところです。
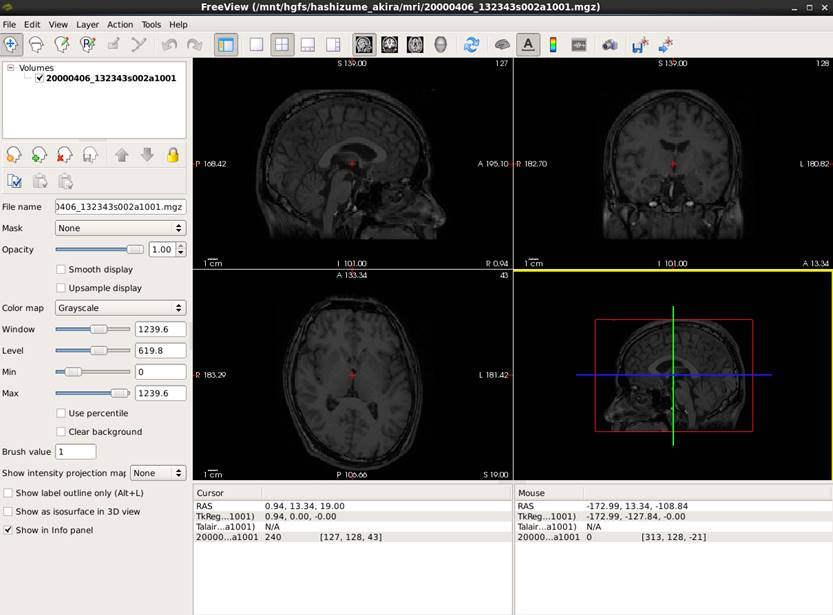
実は変換しなくてもni.gzファイルは直接読めます。
tkmeditは直接は開けません。
[centos@localhost
surf]$ tkmdit hashizume_akira 20000406_132343s002a1001.mgz
tkmdit:
Command not found.
一旦他のファイルで開いてFile→Load Main Volume…で開きます。
[centos@localhost
surf]$ tkmdit hashizume_akira orig.mgz
これで読み込めました。
こちらもfreeview同様、わざわざmgzファイルに変換しなくてもnii.gzファイルは直接読めます。
FreeSurferのsurfファイルをGIfTIファイルに変換
mris_convert
lh.pial lh.pial.gii
mris_convert
rh.pial rh.pial.gii
四面Anatomistで元画像と一緒に表示してみます。
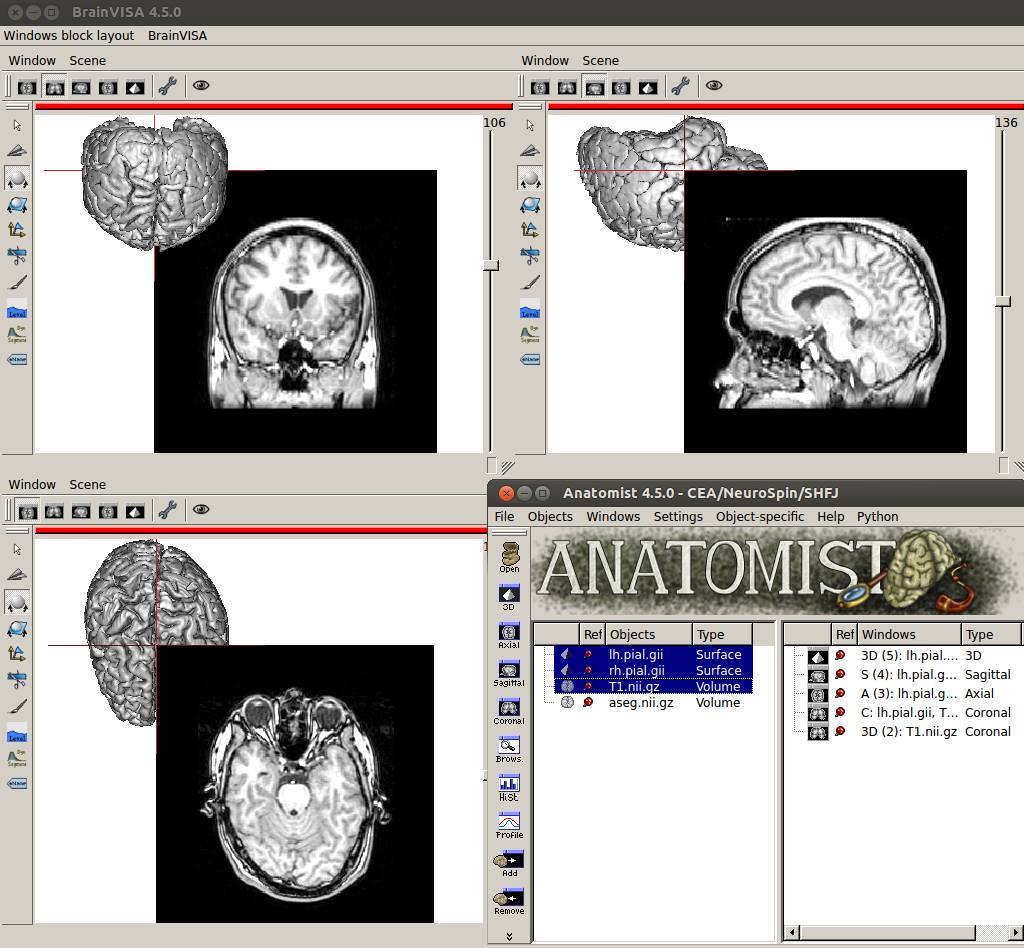
座標はあってません。
MATLABとGIfTI Library for MATLABを使って座標合わせを行います。
GIfTI Library for MATLABはネット上で公開されているツールです。
以下はMATLABのコードです。
addpath('c:/ToBeInstalled/MATLAB/gifti-1.6/');%
GIfTI Library for MATLABにパスを通す
pathname='c:/Users/akira/';%
読み書きファイルの場所
filename={'lh.pial.gii','rh.pial.gii'};%
mris_convertでGIfTI化したファイル
X=[-1,0,0,128;0,-1,0,128;0,0,-1,128;0,0,0,1]';%
座標変換行列
for n=1:length(filename)
g=gifti([pathname,filename{n}]);
P=g.vertices;
P(:,4)=1;
P=P*X;
gg=g;
gg.vertices=P(:,1:3);
gg.faces=g.faces(:,[3,2,1]);% [1,2,3]では三角メッシュの表示が変
save(gg,[pathname,filename{n}(1:end-4),'00.gii'],'Base64Binary');%
ファイル名を00.giiとする
end
これで座標が合いました。
GIfTIファイルをFreeSurferのsurfファイル変換
mris_convertを使えばFreeSurferのsurfファイルになりますが、freeviewだと変換しなくてもそのまま読めます。。
但し、座標変換する必要があります。
MATLABのコードは以下の通りです。面倒ですが、freeviewとtksurferで座標変換部分が異なっていて、それぞれ書き換える必要があります。
clear;
addpath('c:/ToBeInstalled/MATLAB/gifti-1.6/');%
GIfTI Library for MATLABにパスを通す
pathname='c:/Users/akira/';%
読み書きファイルの場所
filename='20000406_132343s002a1001.nii.gz';%
元の三次元配列データ
v=niftiread([pathname,filename]);
info=niftiinfo([pathname,filename]);
filename={'20000406_132343s002a1001_Lhemi.gii','20000406_132343s002a1001_Rhemi.gii';};
d=size(v).*info.PixelDimensions/2;%
補正用
% freeview用
X1=[-1,0,0,d(1);
0,1,0,-d(2);
0,0,-1,d(3);
0,0,0,1]';
x1=[1,3,2];
st1='00.gii';
% tksurfer用
X2=[-1,0,0,d(1);
0,-1,0,d(2);
0,0,-1,d(3);
0,0,0,1]';
x2=[1,2,3];
st2='000.gii';
X=X2;x=x2;st=st2;
for n=1:length(filename)
g=gifti([pathname,filename{n}]);
P=g.vertices;
P(:,4)=1;
P=P*X;
gg=[];
gg.mat=eye(4);
gg.faces=g.faces(:,[3,2,1]);% [1,2,3]では三角メッシュの表示が変
gg.vertices=P(:,x);
gg=gifti(gg);
savename=[filename{n}(1:end-4),st];
save(gg,[pathname,savename],'Base64Binary');
end
freeviewで読んでみました。
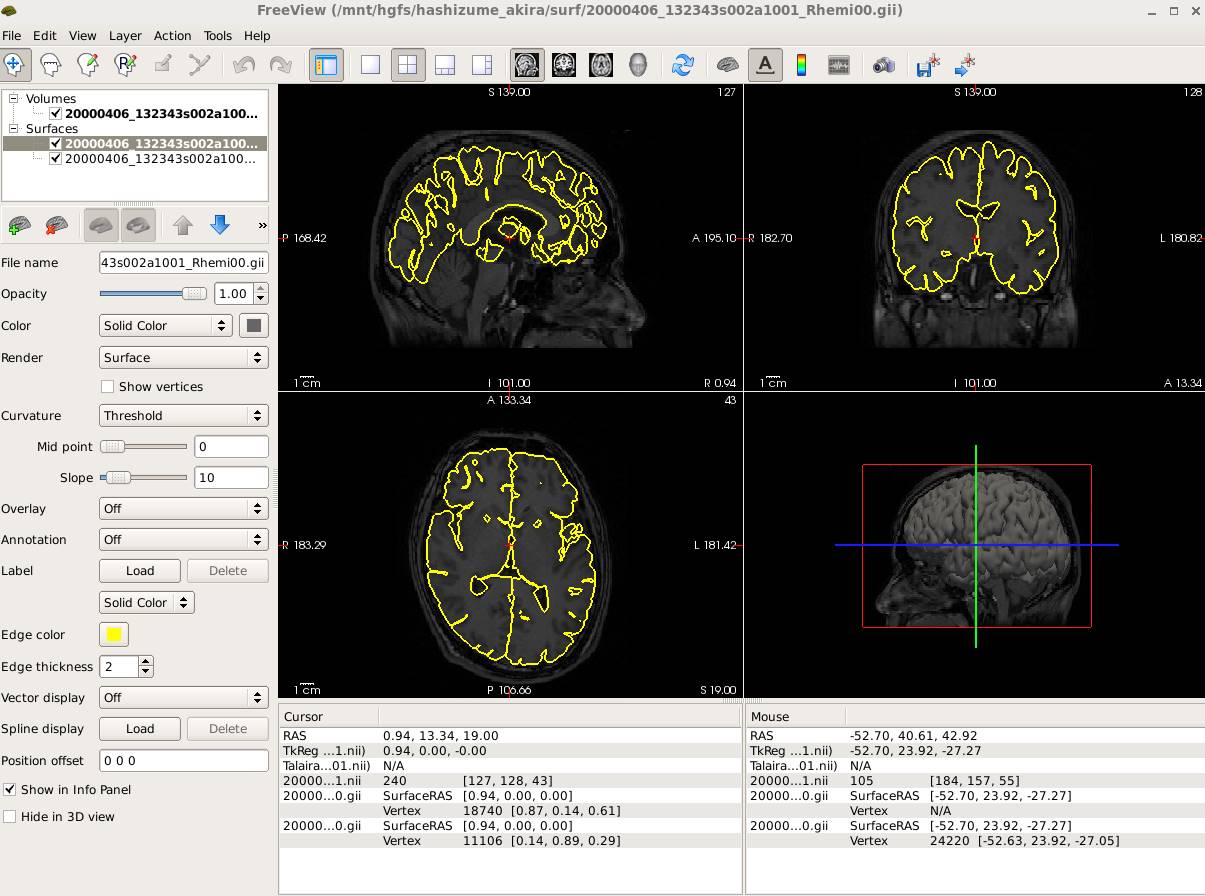
tksurferで読んでみます。mirs_convertでFreeSurfer用のsurfファイルに変換します。接頭語として左右を分けるlh.とかrh.をつけてください。
[centos@localhost
~/Desktop]$ cd ./hashizume_akira/surf
[centos@localhost
surf]$ mris_convert 20000406_132343s002a1001_Lhemi000.gii lh.hemi_pial
Saving
lh.hemi_pial as a surface
ではtksurferで読んでみます。
[centos@localhost
surf]$ tksurfer hashizume_akira lh hemi_pial
subject
is hashizume_akira
hemi is lh
surface
is hemi_pial
surfer:
current subjects dir: /home/centos/Desktop
surfer:
not in "scripts" dir ==> using cwd for session root
surfer:
session root data dir ($session) set to:
surfer:
/mnt/hgfs/hashizume_akira/surf
checking
for nofix files in 'hemi_pial'
Reading
image info (/home/centos/Desktop/hashizume_akira)
Reading
/home/centos/Desktop/hashizume_akira/mri/orig.mgz
surfer:
Reading header info from /home/centos/Desktop/hashizume_akira/mri/orig.mgz
surfer:
vertices=57535, faces=115066
mrisReadTriangleFile(/home/centos/Desktop/hashizume_akira/surf/lh.orig):
surface doesn't match /home/centos/Desktop/hashizume_akira/surf/lh.hemi_pial
mrisReadTriangleFile
failed.
MRISreadOriginalProperties:
could not read surface file /home/centos/Desktop/hashizume_akira/surf/lh.orig
surfer:
single buffered window
surfer:
tkoInitWindow(hashizume_akira)
setting
percentile thresholds (-1.00, 0.00, 0.00)
surfer:
using interface /usr/local/freesurfer/tktools/tksurfer.tcl
Reading
/usr/local/freesurfer/tktools/tkm_common.tcl
Reading
/usr/local/freesurfer/tktools/tkm_wrappers.tcl
Reading
/usr/local/freesurfer/lib/tcl/fsgdfPlot.tcl
Reading
/usr/local/freesurfer/tktools/tkUtils.tcl
Successfully
parsed tksurfer.tcl
reading
white matter vertex locations...
mrisReadTriangleFile(/home/centos/Desktop/hashizume_akira/surf/lh.white):
surface doesn't match /home/centos/Desktop/hashizume_akira/surf/lh.hemi_pial
mrisReadTriangleFile
failed.
%
エラーは出ますが、とりあえず読めます。
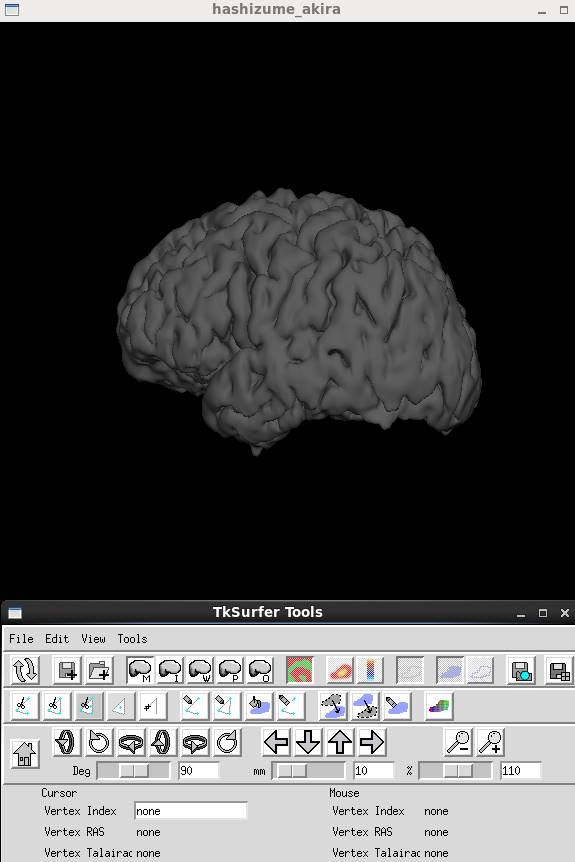
一旦tksurferで
tksurfer 被検者名 lh pial
とかで起動後に表面を選択するという方法であればGIfTIファイルを直接読み込めます。
File→Load Surface…を選択し、そこでgiiファイルかmris_convertで変換したファイルを読みます。
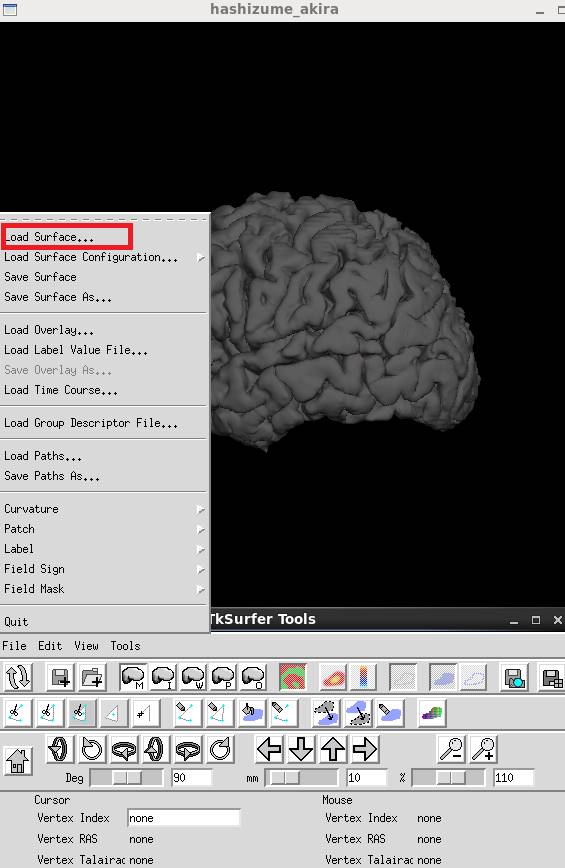
GIfTIファイルが読みこめました。